Enfoque de descomposición natural
(Existen posiciones disponibles para tesis doctorales y de licenciatura en este proyecto)
En la literatura científica existen diversos métodos para identificar módulos en redes complejas, pero ninguno de estos métodos explota el conocimiento biológico que tenemos sobre las redes. Por el contrario, estos métodos están basados en procesos de optimización que pueden encontrar soluciones subóptimas o se basan en parámetros no objetivos sin ningún significado biológico.
Para resolver estas limitaciones, nosotros desarrollamos el enfoque de descomposición natural (EDN). El EDN define criterios objetivos para identificar los sistemas y elementos a nivel de sistemas en una red regulatoria, así como reglas para revelar su arquitectura funcional mediante descomposición controlada (Figura 1). Este enfoque motivado biológicamente deriva matemáticamente la arquitectura y elementos a nivel de sistemas a partir de las propiedades estructurales globales de una red regulatoria dada. Este está basado en dos premisas biológicas:
- Un módulo es un conjunto de genes cooperando para realizar una función fisiológica particular, confiriendo así diferentes rasgos fenotípicos a la célula.
- Dado el efecto pleiotrópico de los reguladores globales, estos no deben pertenecer a módulos sino por el contrario coordinarlos en respuesta a señales ambientales de interés general.
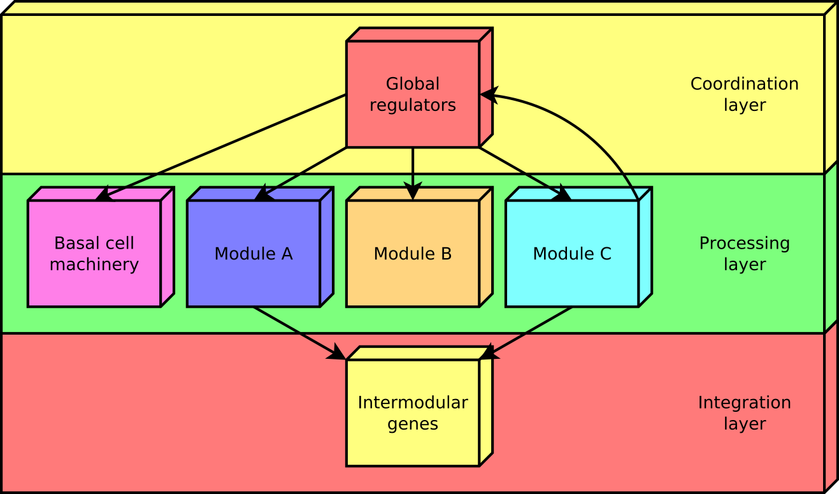
De acuerdo con este enfoque, cada gen en una red regulatoria se predice que pertenece a uno de cuatro posibles clases de elementos a nivel de sistemas, los cuales se interrelacionan en una jerarquía, no piramidal, de tres niveles moldeando la arquitectural funcional como sigue: (1) Los reguladores globales son responsables por coordinar tanto la (2) maquinaria basal celular, compuesta de genes regulados exclusivamente de forma global (EGRGs), y (3) módulos localmente autónomos (conformados por los genes modulares), mientras que (4) los genes intermodulares integran, a nivel promotor, respuestas de los módulos fisiológicamente disparatadas permitiendo un procesamiento combinatorio de las señales ambientales (Figura 2). Este último un nuevo elemento nunca antes descrito en las redes de regulación, el cual fue identificado por primera vez mediante el EDN.
Existen un par de ventajas resultantes de desarrollar un método ad-hoc basado en conocimiento biológico: (1) Los reguladores globales (hubs) no pertenecen a ningún módulo. Esto es biológicamente importante porque ellos no están relacionados con alguna función fisiológica en particular. (2) Identificamos que existe traslape entre módulos y que éste es mediado por los genes intermodulares. Ninguna de estas características clave existentes en redes regulatorias bacterianas hubiera sido identificadas con alguno de los métodos comúnmente empleados para identificar comunidades en redes complejas. Finalmente, el EDN no sólo brinda un método para identificar los elementos a nivel de sistemas moldeando las redes regulatorias bacterianas, sino que este además abre el caminio hacia una teoría de los principios que gobiernan la organización y evolución de estas redes de control celular.
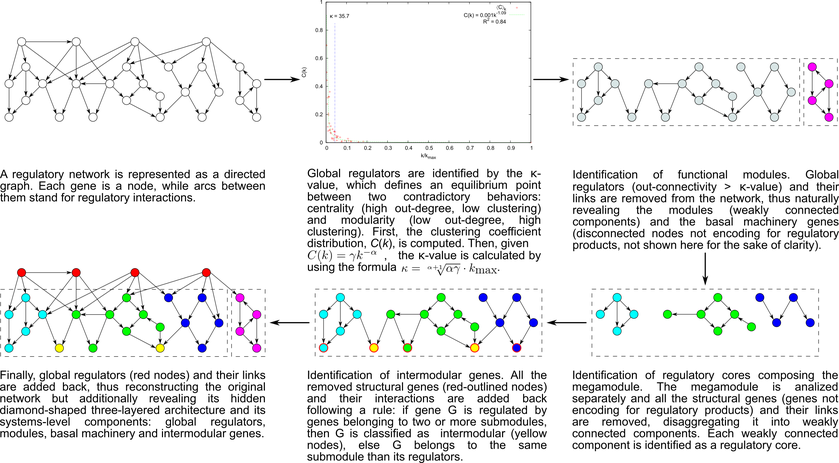
Figura 1. El enfoque de descomposición natural.
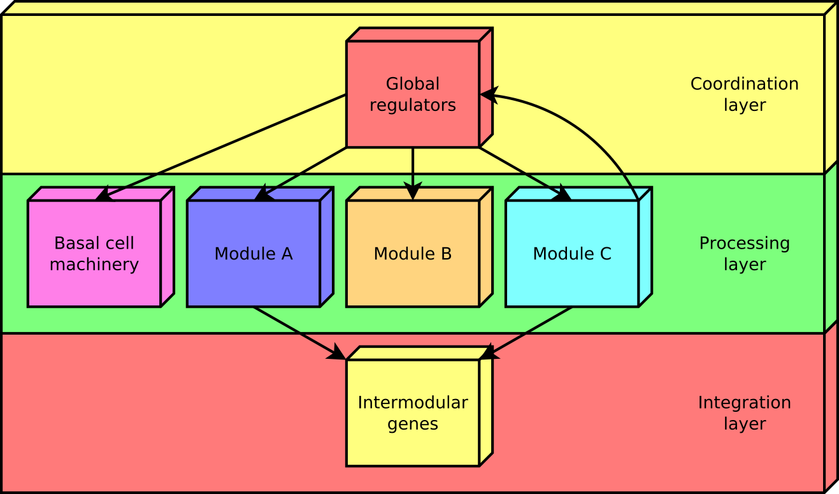
Figura 2. La arquitectura funcional develada por el EDN es una jerarquía de tres niveles en forma de diamante presentando retroalimentación entre los niveles de procesamiento y coordinación, la cual es moldeada por cuatro clases de elementos a nivel de sistemas: reguladores globales, módulos localmente autónomos, maquinaria basal, y genes intermodulares.
- Freyre-Gonzalez, J.A.*, Escorcia-Rodríguez, J.M., Gutiérrez-Mondragón, L.F., Martí-Vértiz, J. Torres-Franco C.N., Zorro-Aranda, A. System principles governing the organization, architecture, dynamics, and evolution of gene regulatory networks. Frontiers in Bioengineering and Biotechnology 10:888732 (2022) doi:10.3389/fbioe.2022.888732
- Escorcia-Rodríguez, J.M., Tauch, A., Freyre-González, J.A.* Corynebacterium glutamicum regulation beyond transcription: Organizing principles and reconstruction of an extended regulatory network incorporating regulations mediated by small RNA and protein-protein interactions. Microorganisms 9(7):1395 (2021) doi:10.3390/microorganisms9071395
- Escorcia-Rodríguez, J.M., Tauch, A., Freyre-González, J.A.* Abasy Atlas v2.2: The most comprehensive and up-to-date inventory of meta-curated, historical, bacterial regulatory networks, their completeness and system-level characterization. Computational and Structural Biotechnology Journal 18:1228-1237 (2020) doi:10.1016/j.csbj.2020.05.015
- Freyre-González, J.A.*, Tauch, A. Functional architecture and global properties of the Corynebacterium glutamicum regulatory network: novel insights from a dataset with a high genomic coverage. Journal of Biotechnology 257C:199-210 (2017) doi:10.1016/j.jbiotec.2016.10.025
- Ibarra-Arellano, M.A., Campos-González, A.I., Treviño-Quintanilla, L.G., Tauch, A., Freyre-González, J.A.* Abasy Atlas: A comprehensive inventory of systems, global network properties, and systems-level elements across bacteria. Database 2016:baw089 (2016) doi:10.1093/database/baw089
- Freyre-González, J.A.*, Treviño-Quintanilla, L.G., Valtierra-Gutiérrez, I., Gutierrez-Ríos, R.M., Alonso-Pavón, J.A. Prokaryotic regulatory systems biology: Common principles governing the functional architectures of Bacillus subtilis and Escherichia coli unveiled by the natural decomposition approach. Journal of Biotechnology 161(3):278-286 (2012) doi:10.1016/j.jbiotec.2012.03.028
- Freyre-Gonzalez, J.A.* and Trevino-Quintanilla, L.G. Analyzing regulatory networks in bacteria. Nature Education 3(9):24 (2010) http://www.nature.com/scitable/topicpage/analyzing-regulatory-networks-in-bacteria-14426192
- Freyre-González, J.A.*, Alonso-Pavón, J.A., Treviño-Quintanilla, L.G., and Collado-Vides, J. Functional architecture of Escherichia coli: new insights provided by a natural decomposition approach. Genome Biology 9(10):R154 (2008) doi:10.1186/gb-2008-9-10-r154